Tabla de enlaces
Resumen e I. Introducción
-
Materiales y métodos
2.1. Aprendizaje de instancias múltiples
2.2. Arquitecturas de modelos
-
Resultados
3.1. Métodos de entrenamiento
3.2. Conjuntos de datos
3.3. Tubería de preprocesamiento de WSI
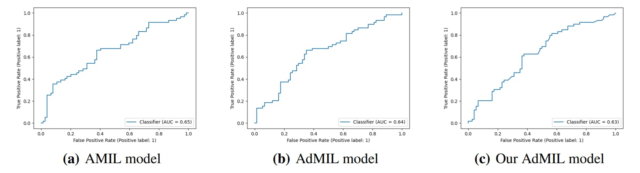
3.4. Resultados de clasificación y detección de ROI
-
Discusión
4.1. Tarea de detección de tumores
4.2. Tarea de detección de mutaciones genéticas
-
Conclusiones
-
Expresiones de gratitud
-
Declaración y referencias del autor
5. Conclusiones
En este trabajo, estudiamos el rendimiento de los marcos de aprendizaje de múltiples instancias (MIL) con mecanismos de atención para la clasificación WSI y la tinción digital de tumores de mama. Exploramos dos enfoques distintos que identifican diferentes representantes de morfología para los clasificadores de parche (mosaico). Estos dos marcos se usaron en una aplicación débilmente supervisada para la detección de tumores y la detección de mutación TP53 en el carcinoma de carcinoma de mama y el carcinoma de células escamosas. Descubrimos que period mucho más fácil identificar regiones de interés (ROI) que reconocen tumor frente a no tumor, incluso a baja resolución (AUC> 0.95), que clasificar TP53 Mutatado frente a no mutado (AUC <0.71). La observación de que las resoluciones más altas (20X) funcionaron mejor para identificar el ROI para la mutación no fue una sorpresa por sí misma, pero las oportunidades para plantear la hipótesis de interpretaciones morfológicas surgieron como el resultado principal del trabajo reportado aquí. Del mismo modo, cuando exploramos nuevas modificaciones del método establecido de aprendizaje de múltiples instancias al alterar su capa de atención authentic, el resultado más interesante no fue la precisión mejorada, sino la atención que el modelo puso en diferentes características morfológicas (es decir, "pintura de diapositivas"). Específicamente, los resultados descritos en las Figuras 3, 4 y 5 ilustran las oportunidades para la exploración interactiva de las morfologías recurrentes para su papel en la etiología del cáncer.
6. Agradecimientos
Este trabajo fue financiado en parte por el Programa de Investigación Intramural del Instituto Nacional del Cáncer (NCI) CAS#10901 (epishere), el Fondo de Recuperación y Resiliencia hacia el Centro de AI responsable (Ref. C628696807-00454142) y el financiamiento de la Fundación para la Ciencia y la Tecnología (FCT) para Inescid (Ref. UIDB/50021/2020).
El código de código abierto utilizado en este proyecto está disponible públicamente en https://github.com/timafonso/wsi_mil_roi
7. Declaración del autor
No hay conflicto de intereses que informar.
Referencias
[1] Bahdanau, D., Cho, KH, Bengio, Y., 2015. Traducción del automóvil neuronal aprendiendo conjuntamente a alinearse y traducir. Tercera Conferencia Internacional sobre Representaciones de Aprendizaje, ICLR 2015 – Actas de la pista de la conferencia, 1-15.
[2] Dietterich, TG, Lathrop, RH, Lozano-Pérez, T., 1997. Resolviendo el problema de instancia múltiple con rectángulos de eje paralelo. Inteligencia synthetic 89, 31–71.
[3] Dimitriou, N., Arandjelović, O., Caie, PD, 2019. Aprendizaje profundo para análisis de imágenes de diapositivas completas: una visión normal. Frontiers in Medication 6, 1–7.
[4] Guo, B., Li, X., Yang, M., Zhang, H., Xu, XS, 2023. Un algoritmo de aprendizaje de instancia múltiple robusta y liviana para predecir alteraciones genéticas. Imágenes médicas computarizadas y gráficos 105, 102189.
[5] Hanna, MG, Parwani, A., Sirintrapun, SJ, 2020. Imágenes de diapositivas enteras: tecnología y aplicaciones. Avances en Patología Anatómica 27, 251–259.
[6] Hou, W., Yu, L., Lin, C., Huang, H., Yu, R., Qin, J., Wang, L., 2022. H2-Mil: Explorando la representación jerárquica con el aprendizaje de múltiples instancias heterogéneas para el análisis de imágenes de diapositivas completa. Actas de la Conferencia AAAI sobre Inteligencia Synthetic 36, 933–941.
[7] Ilse, M., Tomczak, J., Welling, M., 2018. El aprendizaje de instancias múltiples profundas basadas en la atención, en: Conferencia internacional sobre aprendizaje automático, pp. 2127–2136.
[8] Javed, SA, Juyal, D., Padigela, H., Taylor-Weiner, A., Yu, L., Prakash, A., 2022. Aditivo MIL: aprendizaje de instancias múltiples intrínsecamente interpretables para la patología. Avances en los sistemas de procesamiento de información neural 35, 20689–20702.
[9] Khened, M., Kori, A., Rajkumar, H., Krishnamurthi, G., Srinivasan, B., 2021. Un marco de aprendizaje profundo generalizado para la segmentación y el análisis de imágenes de plana completa. Informes científicos 11, 1–14.
[10] Konstantinov, AV, Utkin, LV, 2022. Aprendizaje de instancias múltiples de atención múltiple. Computación y aplicaciones neurales, 1–23.
[11] Li, B., Li, Y., Eliceiri, KW, 2021. Pink de aprendizaje de instancias de múltiples instancias duales para una clasificación de imagen de diapositivas completa con aprendizaje contrastante auto-supervisado. Actas de la IEEE Pc Society Conferencia sobre visión por computadora y reconocimiento de patrones, 14313–14323.
[12] Li, Z., Yuan, L., Xu, H., Cheng, R., Wen, X., 2020. Aprendizaje profundo de múltiples instancias con autoatención inducida para la clasificación de imágenes médicas. Actas: 2020 Conferencia Internacional IEEE sobre bioinformática y biomedicina, BIBM 2020, 446–450.
[13] Lu, M., Pan, Y., Nie, D., Liu, F., Shi, F., Xia, Y., Shen, D., 2021a. Sonrisa: aprendizaje contrastante de instancia múltiple basada en atención escasa para la clasificación de subtipo de glioma utilizando imágenes patológicas, en: Miccai Workshop on Computational Pathology, pp. 159-169.
[14] Lu, My, Williamson, DF, Chen, Ty, Chen, RJ, Barbieri, M., Mahmood, F., 2021b. Patología computacional de eficiencia de datos y débilmente supervisada en imágenes de plana completa. Ingeniería biomédica de la naturaleza 5, 555–570.
[15] Nayak, N., Chang, H., Borowsky, A., Spellman, P., Parvin, B., 2013. Clasificación de la histopatología tumoral a través del aprendizaje de características dispersas, en: 2013 IEEE 10º Simposio internacional sobre imágenes biomédicas, págs. 410–413.
[16] Otsu, N., 1979. Un método de selección de umbral de histogramas de nivel de grises. Transacciones IEEE en sistemas, hombre y Cybernetics 9, 62–66.
[17] Reisenbüchler, D., Wagner, SJ, Boxberg, M., Peng, T., 2022. Transformador basado en gráficos de atención native para la predicción de alteración genética de múltiples objetivos, en: Conferencia internacional sobre informática de imágenes médicas e intervención asistida por computadora, Springer. pp. 377–386.
[18] Riasatian, A., Babaie, M., Maleki, D., Kalra, S., Valipour, M., Hemati, S., Zaveri, M., Safarpoor, A., Shafiei, S., Afshari, M., Rasoolijaberi, M., Sikaroudi, M., Adnan, M., Shah, S., Choi, C., Damaskinos, S., Campbell, CJ, Diamandis, P., Pantanowitz, L., Kashani, H., Ghodsi, A., Tizhoosh, H., 2021. Autorización y entrenamiento de Densenet para la representación de imágenes de histopatología utilizando toboganes de diagnóstico TCGA. Análisis de imágenes médicas 70, 102032.
[19] Rymarczyk, D., Borowa, A., Tabor, J., Zielinski, B., 2021. Autoatención del núcleo para la clasificación de imágenes débilmente supervisada utilizando un aprendizaje profundo de instancias múltiples. Actas – Conferencia de invierno 2021 IEEE sobre aplicaciones de visión por computadora, WACV 2021, 1720–1729.
[20] Shao, Z., Bian, H., Chen, Y., Wang, Y., Zhang, J., Ji, X., et al., 2021. Transmila: aprendizaje de instancias múltiples correlacionadas basadas en transformador para la clasificación de imagen de diapositiva completa. Avances en los sistemas de procesamiento de información neural 34, 2136–2147.
[21] Sheikh, TS, Kim, JY, Shim, J., Cho, M., 2022. Aprendizaje sin supervisión basado en múltiples descriptores para el diagnóstico de WSIS. Diagnóstico 12, 1480.
[22] Tomczak, Okay., Czerwińska, P., Wiznerowicz, M., 2015. El genoma del cáncer Atlas (TCGA): una fuente inconmensurable de conocimiento. Wspolczesna Onkologia 1A, A68 – A77.
[23] Wang, X., Chen, H., Gan, C., Lin, H., Dou, Q., Tsougenis, E., Huang, Q., Cai, M., Heng, PA, 2020. El aprendizaje profundo débilmente supervisado para el análisis de imagen de cáncer de pulmón de tobogán entero. Transacciones IEEE en Cybernetics 50, 3950–3962.
[24] Wang, X., Zou, C., Zhang, Y., Li, X., Wang, C., Ke, F., Chen, J., Wang, W., Wang, D., Xu, X., et al., 2021. Predicción de la mutación del gen BRCA en el cáncer de mama basado en el aprendizaje profundo e imágenes de histopatología. Frontiers in Genetics 12, 661109.
[25] Yao, J., Zhu, X., Jonnagaddala, J., Hawkins, N., Huang, J., 2020. Predicción de supervivencia de cáncer basada en imágenes de deslizamiento entero utilizando redes de aprendizaje de múltiples instancias múltiples guiadas por la atención. Análisis de imágenes médicas 65.
[26] Zhao, L., Yuan, L., Hao, Okay., Wen, X., 2022. Aprendizaje generalizado de múltiples instancias basadas en la atención. Sistemas multimedia, 1-13.
[27] Zhu, X., Yao, J., Zhu, F., Huang, J., 2017. WSISA: haciendo una predicción de supervivencia a partir de imágenes histopatológicas de diapositivas completas. Actas – 30ª Conferencia IEEE sobre visión por computadora y reconocimiento de patrones, CVPR 2017 2017 -Janua, 6855–6863.
Autores:
(1) Martim Afonso, Instituto Superior Técnico, Universidade de Lisboa, Av. Rovisco Pais, Lisboa, 1049-001, Portugal;
(2) Praphulla MS Bhawsar, División de Epidemiología y Genética del Cáncer, Instituto Nacional del Cáncer, Institutos Nacionales de Salud, Bethesda, 20850, Maryland, EE. UU.;
(3) Monjoy Saha, División de Epidemiología y Genética del Cáncer, Instituto Nacional del Cáncer, Institutos Nacionales de Salud, Bethesda, 20850, Maryland, EE. UU.;
(4) Jonas S. Almeida, División de Epidemiología y Genética del Cáncer, Instituto Nacional del Cáncer, Institutos Nacionales de Salud, Bethesda, 20850, Maryland, EE. UU.;
(5) Arlindo L. Oliveira, Instituto Superior Técnico, Universidade de Lisboa, Av. Rovisco Pais, Lisboa, 1049-001, Portugal e Inesc-ID, R. Alves Redol 9, Lisboa, 1000-029, Portugal.
Este documento es